新科技速遞
生物的蛋白質由氨基酸串組成,摺疊成3D形狀,決定了蛋白質在細胞中功能,多年來研究人員致力重現高解像3D蛋白質結構,以促進研發藥物設計和生產。
雖然基因研究突飛猛進,但基因所以重要,全因帶有生產蛋白質密碼,生物利用密碼轉變成蛋白質,而在細胞內,蛋白質才是真正功能的執行者。
蛋白質3D結構無法直接觀察,研究人員一直使用X射線晶體學(X-ray Crystallography)或核磁共振儀 (NMR)等技術測定蛋白質3D結構,但進度相當緩慢。
近年,低溫電子顯微鏡(Cryo-TEM)分析蛋白質結構的效率大大提高;去年香港大學獲李嘉誠捐贈了1億港元,成立「低溫電子顯微設施研究中心」,顯微鏡可凍結移動中的微細生物分子,以原子尺度描繪,加快分析蛋白3D結構,推動本港藥物研發工業。
AI脫穎而出
上述數種方法既費時又昂貴,部分蛋白質不適合以上述方法分析;X射線晶體學數個月才測定一種結構,Cryo-TEM設備極之昂貴,須高性能運算配合,安裝和配置設備,也成本不菲。
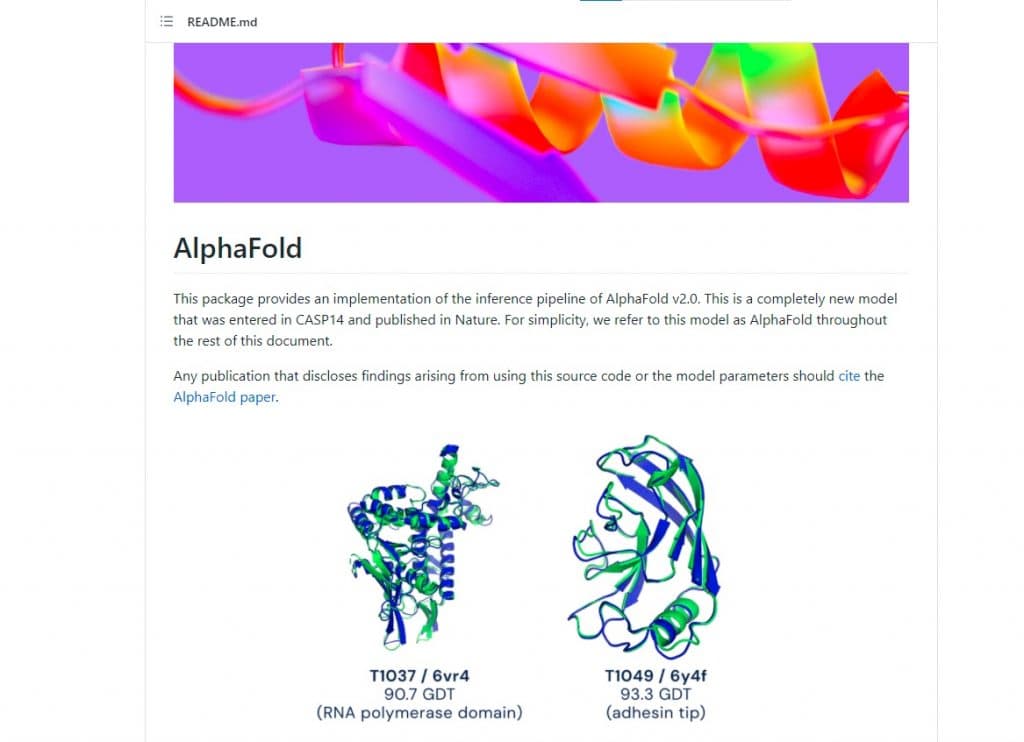
去年12 月,兩年一度舉行的「國際蛋白質結構預測比賽」CASP,英國初創DeepMind以AlphaFold的AI模型,預測出多個蛋白質的 3D 結構,擊敗所有參賽者,準確性與實驗結果匹配,準確度前所未見,一時技驚四座,頓成為生物訊息學最矚目的科技。
DeepMind 開發的AlphaFold 模型,只根據蛋白質氨基酸的序列,就預測出3D 的結構,破解蛋白質折疊困擾生物學的主要難題;AlphaFold以多序列對齊,進行深度學習演算開發AI模型,結合蛋白質結構物理和生物學,開啟出生物信息學的新時代。
DeepMind以AI利用蛋白的基因序列,凖確推測蛋白的結構,不止震驚了學術界,也導致研究上百花齊放,爭相開發AI的模型。

開源模型速度高
上周,《自然》期刊的報導,DeepMind公佈名為AlphaFold 2深度學習神經網的開源版本,與去年參加CASP的比賽版本,已經脫胎換骨,模型仍以Python語言開發,但速度更快。DeepMind在《自然》期刊上,發表《以AlphaFold精凖預測蛋白結構》的論文。
同一天,受AlphaFold 2模型的啟發,另一組學術研究人員也公佈了另一個蛋白預測模型RoseTTaFold,發表論文和開源代碼,預測能力亦媲美AlphaFold 2。
如何預測蛋白質3D 結構,困擾生物學長達50年之久。AlphaFold 2和RoseTTaFold開源AI模型,代表學術界以上述開源工具,理解細胞蛋白質功能,一同改進AI模型凖確度。
以往DeepMind對正進行AI研究,往往是諱莫如深,學術界遂自行研究,美國西雅圖華盛頓大學生物化學家David Baker團隊,摹仿AlphaFold 2開發AI預測蛋白質3D 結構開發,顯示學術界亦掌握AI預測蛋白結構的能力。
雖然RoseTTaFold功能毫不遜色,凖確度則與AlphaFold 仍有落差 ,原因可能工程技術仍有差距,而不是分析方法較差。
DeepMind改進AlphaFold 2演算速度,較CASP 比賽之際,AlphaFold花上數天才預測簡單蛋白結構,開源版本分析速度提高16倍,往往數小時就可獲結果。
AlphaFold 2和RoseTTaFold源碼已上載到GitHub,AlphaFold 2以容器開發,更易安裝和設定,亦與研究機構合作推出新藥;包括日内瓦「被忽視疾病藥物研發」(Drugs for Neglected Diseases)組織合作預測蛋白結構,加速研發罕見病新藥。
透過結構生物學研究蛋白質結構與功能之間的關係,可開發出臨床小分子藥物。過去20年,生物技術療法推陳出新,小分子藥物市場萎縮,AI可加快發現生物標記和標靶,有機會重振小分子藥物的市場。
李嘉誠旗下的維港投資亦是DeepMind早期投資者之一,其後出售予Google。